Desarrollados robots moleculares capaces de nadar coordinadamente
Unos investigadores de las universidades de Hokkaido y Kansai, ambas en Japón, han desarrollado robots moleculares asistidos por ADN que forman enjambres de forma autónoma en respuesta a señales químicas y físicas. El avance abre un camino hacia el desarrollo de futuras nanomáquinas.
Cada uno de estos robots mide 25 nanómetros de diámetro y 5 micrómetros de longitud, y es capaz de exhibir el comportamiento de movimiento en enjambre, típico de animales como peces, hormigas y pájaros.
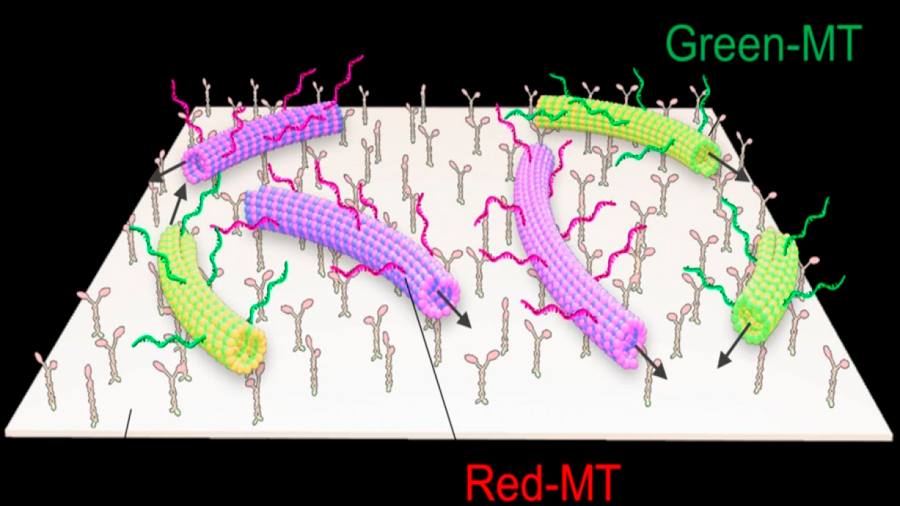
El sistema molecular construido por el equipo de Akira Kakugo está compuesto por los tres componentes esenciales de un robot: sensores, procesadores de información y mecanismos para ejecutar movimientos y desplazarse. Kakugo y sus colegas utilizaron unas proteínas celulares llamadas microtúbulos y kinesinas para ejecutar los movimientos, así como ADN para procesar información. Los microtúbulos son proteínas filamentosas que sirven como raíles en el sistema de transporte celular, mientras que las kinesinas son proteínas motoras que circulan por los raíles gracias al consumo de energía química obtenida de la hidrólisis del trifosfato de adenosina (ATP). El equipo adoptó una estrategia contraria y construyó un sistema en el que los microtúbulos se mueven libremente sobre una superficie recubierta de kinesinas.
Un gran reto para la robótica de enjambre es la construcción de una gran cantidad de robots individuales capaces de un autoensamblaje programable. El equipo afrontó este problema introduciendo moléculas de ADN de un tipo especial.
Inicialmente, cinco millones de microtúbulos se movían sin interaccionar entre sí. Los investigadores añadieron entonces las citadas moléculas de ADN, y los microtúbulos empezaron a ensamblarse y a formar grandes enjambres. Cuando se añadió otro ADN especial de una única hebra, programado para disolver los enjambres de microtúbulos, estos se disociaron pronto. Ello demostró que la reunión en enjambre de una gran cantidad de microtúbulos se puede regular de forma reversible introduciendo la señal adecuada de ADN en el sistema.
Kakugo y sus colegas añadieron además un sensor fotosensible al sistema, concretamente azobenceno unido a moléculas de ADN. Se valieron de la isomerización del azobenceno, que sucede de forma reversible en respuesta a la irradiación de luz visible o ultravioleta, para activar o desactivar la interacción entre las moléculas de ADN. Esto permitió que la fotoirradiación indujera la conmutación entre los estados solitario y en enjambre de los microtúbulos. El equipo también demostró que los enjambres de microtúbulos se mueven con un movimiento de traslación o bien de rotación dependiendo de la rigidez de los microtúbulos.
Estamos en definitiva ante la primera evidencia de que el comportamiento en enjambre de los robots moleculares se puede programar mediante computación de ADN. El sistema actúa como un ordenador básico llevando a cabo funciones lógicas sencillas, como las AND (y) u OR (o), lo cual desemboca en la formación de diversas estructuras y la ejecución de movimientos complejos. Se espera que en el futuro tal sistema contribuya a desarrollar una nueva generación de músculos artificiales, así como a construir nanomáquinas avanzadas, tal como aventura Kakugo.
Fuente: noticiasdelaciencia.com