Luz visible contra un enemigo invisible
Hilda Mercado Uribe
Se doctoró en la Universidad de Guanajuato y actualmente es investigadora de tiempo completo en la Unidad Monterrey del Cinvestav. Sus líneas de investigación contemplan el estudio de hidratación/deshidratación de biomoléculas y bacterias, así como efectos de luz visible en células. Es nivel II del SNI.
Durante el último siglo se ha publicado un gran número de trabajos científicos sobre el éxito obtenido con el uso de agentes antimicrobianos; es decir, fármacos utilizados para curar infecciones provocadas por bacterias, virus, hongos y parásitos. Desafortunadamente, después del éxito, se presenta un hecho natural e inevitable: los microorganismos en cuestión desarrollan resistencia a los fármacos. En el caso particular de las bacterias, este hecho se conoce como resistencia antibiótica y existen varias razones para que ocurra. Una de ellas se debe a que el mecanismo de acción de los antibióticos en la célula es específico. Las drogas se fabrican basándose en el principio de llave-cerradura para que actúen directamente sobre moléculas blanco (targets) en la célula. Las bacterias cuentan con un sistema de proteínas transportadoras encargadas de expulsar al medio circundante sustratos tóxicos para ella, como es el caso de los antibióticos. Estas proteínas llamadas bombas de flujo de salida (efflux pumps) pueden sobre-expresarse en la célula y ocasionar mutaciones en los genes que codifican los sitios blanco en donde se une el antibiótico. Junto con ello, como el mecanismo de replicación celular es rápido, es altamente probable que ocurra una mutación, y si en ese momento un agente antibiótico se encuentra en la célula, ésta desarrollará resistencia al fármaco, y tal manifestación prevalecerá en el proceso de reproducción de la población celular [1].
No solo la resistencia a los antibióticos sino en general la resistencia antimicrobiana, se acentúa por la contribución de diversos factores sociales por todos conocidos: la prescripción inadecuada de los fármacos, el uso inapropiado de los mismos por parte de la población (incluyendo la interrupción del tratamiento médico), la falta de políticas públicas para implementar medidas regulatorias, y el intenso movimiento transcontinental y migratorio en el ámbito mundial, que abona a la propagación de infecciones endémicas; tal como está ocurriendo actualmente con el brote del síndrome respiratorio agudo grave (SARS) causado por el coronavirus SARS-COV-2 [2]. Así, la resistencia antimicrobiana tiene un serio impacto en la salud pública de la población, convirtiéndose en una carga social y económica para los países.
De acuerdo con la Organización Mundial de la Salud (OMS), actualmente unas 700,000 personas mueren en el mundo cada año por enfermedades resistentes a los fármacos. Con este antecedente y un análisis de la situación por regiones, el organismo alertó sobre una crisis sanitaria dentro de pocas décadas, la cual tendría efectos muy negativos si no se lleva a cabo un plan de acción contundente para enfrentar el problema. En este plan se conmina a los países a realizar “inversión y desarrollo de nuevas tecnologías para combatir la resistencia a los agentes antimicrobianos” [3]. Con relación a este punto, desde finales del siglo pasado, diversos grupos de investigación han explorado la aplicación de un método conocido como Inactivación Fotodinámica (PDI, por sus siglas en inglés). Cabe señalar que la PDI se ha utilizado ya con éxito como terapia contra el cáncer, logrando la muerte de diversos tipos de células tumorales (2).
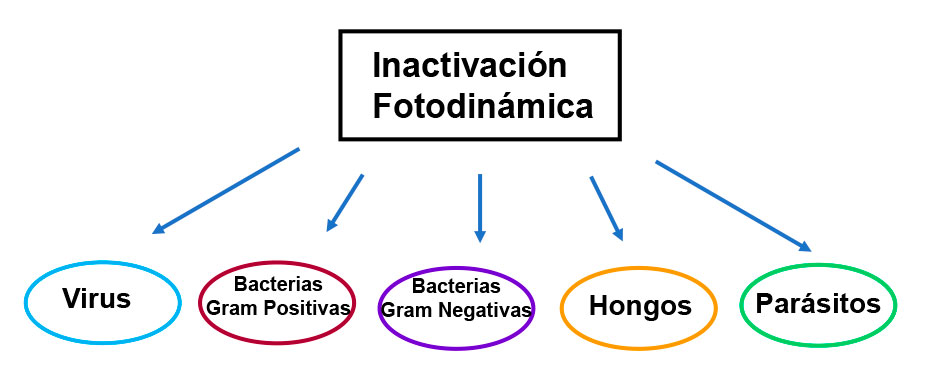
La PDI es considerada como una potente alternativa para inhibir microorganismos que son muy resistentes, ya sea por sus características per se, o por haber desarrollado tal fortaleza ante diferentes drogas (ver Fig. 1). Este método garantiza, además, que no habrá resistencia antimicrobiana en un futuro. Otra ventaja es que después de la irradiación, el fotosensibilizador produce un residuo que no daña al medio ambiente. El procedimiento se basa en el uso de moléculas no-tóxicas y fotosensibles, que al excitarlas con luz ultravioleta (UV) o visible de longitud de onda apropiada, y en presencia de oxígeno molecular, generan especies reactivas de oxígeno (ROS, por sus siglas en inglés). Más adelante ahondaremos en el origen y la relevancia de estas moléculas en la PDI. Por ahora, hablemos en general de los eventos que se van desarrollando durante la aplicación de este método (ver Fig. 2):
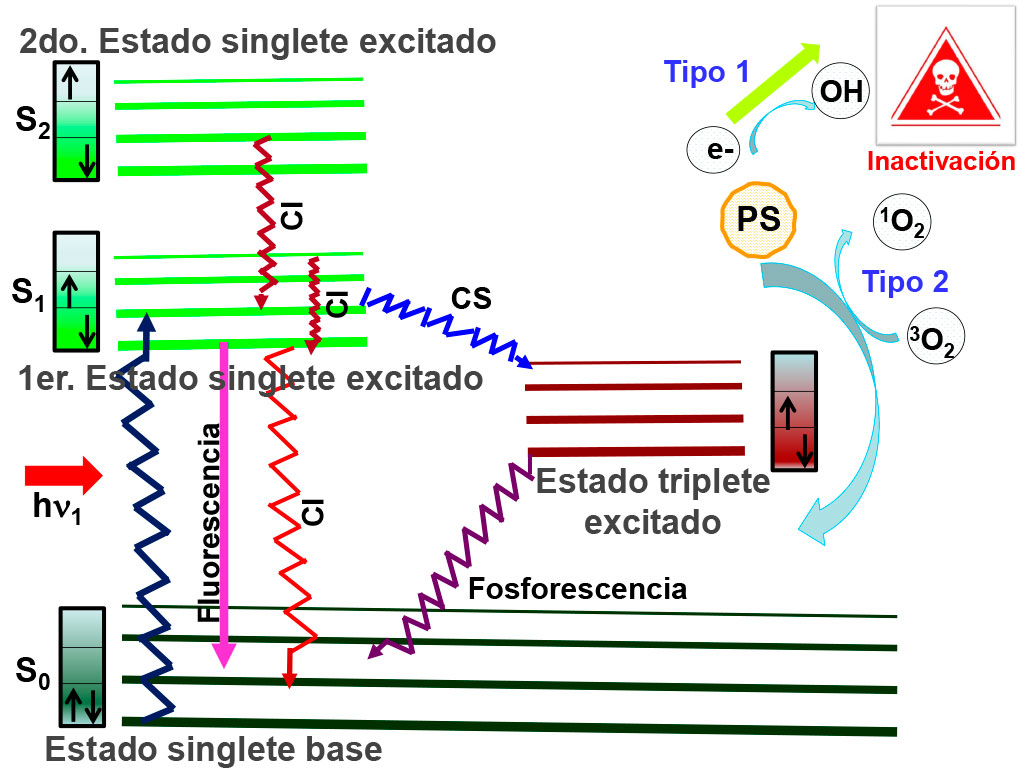
Inicialmente, la molécula fotosensibilizadora (PS) se encuentra como la naturaleza lo dicta, en su estado base o de mínima energía, que en este caso se conoce como estado singlete (S0), caracterizado por tener dos electrones externos apareados (sus números cuánticos de espín son antiparalelos). Cuando el fotosensibilizador es excitado con la longitud de onda a la que éste muestra la mayor absorbancia, esa energía empuja a uno de los electrones a un estado más energético, llamado estado singlete excitado (S1), el cual es más inestable que el S0 y tiene una vida media muy corta (del orden de nanosegundos). La energía suministrada puede ser tal que lleve al PS a un estado aún más energético, como S2, S3, S4,… y así, sucesivamente, perdiendo eventualmente todo ese exceso de energía, y cayendo gradualmente al estado base a través de varios mecanismos. Uno de ellos, por fluorescencia (con emisión de luz) y el otro, por conversión interna (con producción de calor). Un tercer mecanismo de decaimiento es por medio del llamado cruce entre sistemas. En este caso, el PS cae a un estado llamado triplete (T1), que es un estado excitado más estable (menos energético) que el estado S1, con una vida media del orden de microsegundos, debido a que uno de los electrones tiene que rotar su espín en el proceso [4]. A partir de este momento, ocurren sucesos más interesantes. La molécula PS en estado T1 puede decaer hasta el estado base mediante dos posibles trayectorias: la más compleja, el proceso Tipo 1, se lleva a cabo mediante reacciones químicas de oxidación y reducción (las conocidas redox), que conducen a la formación de ROS, principalmente, anión superóxido (O2.-), peróxido de hidrógeno (H2O2) y radical hidroxil (OH·). El superóxido, en particular, no es biológicamente reactivo; sin embargo, cuando se protona, es un detonador de cascadas de ROS. Más trascendental es el decaimiento que se da por la vía del proceso Tipo II. Aquí, el PS transfiere su energía directamente al oxígeno molecular (O2) del medio. Esta molécula es un radical libre per se; es decir, sus dos electrones más externos de cada átomo componente están desapareados, por lo que el O2 tiene un comportamiento muy inquieto, y para subsanar esta falta de compañeros, roba electrones a otras moléculas del medio. Este inusual estado de máxima multiplicidad (más alta disponibilidad) y de más baja energía, se conoce como estado triplete (3O2). El proceso Tipo II ocurre más rápido que el proceso Tipo 1, y da lugar a una nueva molécula, más energética y altamente reactiva conocida como oxígeno singlete (1O2). Ésta es considerada la especie más tóxica generada en PDI. Precisamente, como el 1O2 es extremadamente reactivo, tiene una vida media muy corta (<4 s), y un radio de acción muy limitado (aproximadamente 150 nm). Una vez que el PS libera todo el exceso de energía absorbida proveniente de la luz y cae finalmente al estado base, puede volver a excitarse y generar nuevas ROS.
Los lípidos y las proteínas son los principales componentes de las membranas biológicas, y son también moléculas blanco sobre las que las especies reactivas liberan su energía electrónica. Por ejemplo, las ROS se “pegan” a un lípido o “roban” un hidrógeno de un grupo metileno (-CH2-), iniciando el proceso conocido como peroxidación lipídica. En particular, el radical hidroxil, generado en el proceso Tipo I antes mencionado, es uno de los iniciadores de la peroxidación de ácidos grasos, lipoproteínas y membranas. Aun cuando el radical OH· se genere afuera de la membrana, éste es capaz de afectar a los carbohidratos extrínsecos y a las glicoproteínas superficiales de la célula; así como a los grupos principales de los fosfolípidos [5]. Por su parte, el radical perhidroxilo (OH·), que es una forma protonada del anión superóxido, producido también en proceso Tipo I, logra extraer H· de algunos ácidos grasos poliinsaturados, como el ácido araquidónico y el linoleico, relacionados con procesos inflamatorios e inmunológicos en los mamíferos.
Las especies reactivas también provocan daños en las proteínas; algunos de ellos son reversibles, como la generación de puentes disulfuro, la formación de ácido sulfénico y la destrucción de grupos de Fe-S. Sin embargo, es posible que los daños sean más críticos, tales que la célula no sea capaz de repararlos, como es el caso de la oxidación de las cadenas laterales de los aminoácidos, en donde los OH pueden sustraer H, o bien, el rompimiento de la cadena principal de péptidos.
La pieza principal de la célula, el ADN, puede también ser afectada por oxidación de las polimerasas y enzimas reparadoras. En tanto que en el ARN puede ser alterado el proceso de traducción, lo que implicaría la generación de proteínas defectuosas [5].
Desde hace algunos años, en nuestro grupo de trabajo hemos investigado la inactivación de la bacteria E. coli, aplicando el método PDI. E. coli, por ser del tipo Gram-negativa, es muy resistente a la inactivación. Para lograrlo, se han explorado diversas moléculas fotosensibles: de origen natural, comercial y sintetizadas en nuestro laboratorio, y se ha potenciado su efecto con agentes quelantes que desorganizan la membrana celular [6], permitiendo la internalización del PS. Todos los fotosensibilizadores son excitados con diferentes tipos de radiación, entre ellas, la luz visible.
La inactivación de parásitos es otra de las vertientes de la PDI, y pronto iniciaremos una investigación con estos microorganismos que producen enfermedades endémicas en el sur del país.
Los virus son agentes infecciosos genéticamente flexibles que mutan velozmente, por lo que pueden adquirir resistencia a las drogas con facilidad. Se puede decir que los virus son de dos tipos: los que están rodeados por una envoltura proteica y los que carecen de ella. El virus de la inmunodeficiencia (VIH) y el del herpes son ejemplos de los primeros; en tanto que el del papiloma (HPV) y el adenovirus entran en la segunda categoría. Virus como el HIV-1, HSV-1, HSV-2, el adenovirus, el de la influenza, el del dengue, el enterovirus [7] entre otros, han tenido una respuesta de inactivación favorable aplicando la PDI y determinadas moléculas fotosensibilizadoras. En dichos trabajos, se ha observado que los virus encapsulados son mucho más sensibles a la PDI que los virus que no están encapsulados [8]. Se ha reportado, además, que algunos virus encapsulados como el HIV y el VSV, disminuyeron significativamente su capacidad de fusionarse después de haber sido tratados con PDI e hipericina y rosa de Bengala [9]. La efectividad de la PDI en el caso de los virus está determinada por la estructura y carga molecular de éste, y por el grado de hidrofobicidad de su membrana.
En este contexto, y considerando la situación de la actual crisis sanitaria en el mundo por el virus SARS-CoV-2, valdría la pena retomar con seriedad las recomendaciones de la OMS, y aplicar la PDI como una alternativa para investigar posibles protocolos para combatir este pertinaz enemigo invisible. Dado que el SARS-COV-2 está constituido por lípidos, proteínas y RNA, es probable que sea afectado directa o indirectamente por las ROS generadas con este método.
Referencias
- [1] T. Dai et al., PPT 6, 170-188 (2009).
- [2] M. Manwright et al., Lancet, 17, e49-e55 (2017)
- [3] World Health Organization, Antimicrobial Resistance Global Report on Surveillance 2014), https://www.who.int/antimicrobial-resistance/publications/surveillancereport/en/
- [4] M. Hamblin, Curr Opin Microbiol, 33, 67-73 (2006)
- [5] B. Halliwell and J.M.C. Gutteridge, Free Radicals in Biology & Medicine, Oxford University Press, 2015.
- [6] R. E. W. Hancock, Ann. Rev. Microbial. 38 237-264 (1984).
- [7] L. Costa et al. Viruses, 4, 1034-1074 (2012).
- [8] M. Manwright, 3, 406-411 (2004).
- [9] J. Lenard et al. PNAS, 90, 158-162 (1993).
Fuente: Revista Avance y Perspectiva