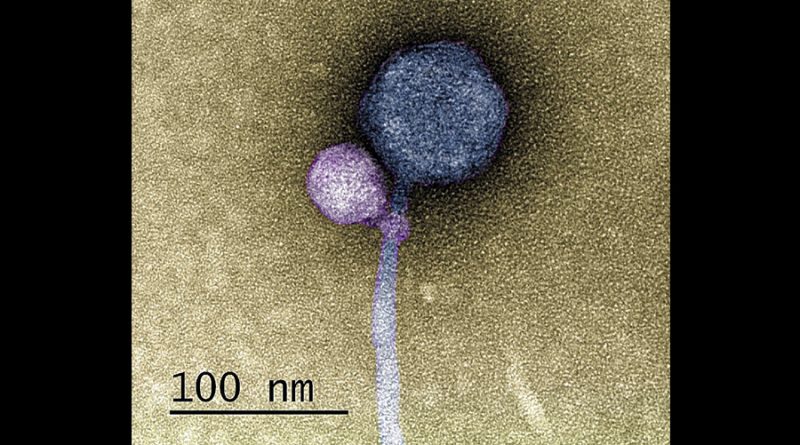
Primera observación de un virus que se une a otro virus
Nadie había visto nunca un virus adherirse a otro virus, hasta que unos resultados anómalos de la secuenciación enviaron a un equipo de la UMBC a un agujero de conejo que condujo a un descubrimiento único en su tipo.
Se sabe que algunos virus, llamados satélites, dependen no sólo de su organismo huésped para completar su ciclo de vida, sino también de otro virus , conocido como “ayudante”, explica Ivan Erill, profesor de ciencias biológicas. El virus satélite necesita ayuda para construir su cápside, una capa protectora que encierra el material genético del virus, o para ayudarlo a replicar su ADN.
Estas relaciones virales requieren que el satélite y el ayudante estén cerca uno del otro al menos temporalmente, pero no se conocen casos de un satélite realmente conectado a un ayudante… hasta ahora.
En un artículo publicado en The ISME Journal , un equipo de la UMBC y colegas de la Universidad de Washington en St. Louis (WashU) describen la primera observación de un bacteriófago satélite (un virus que infecta células bacterianas ) que se adhiere constantemente a un bacteriófago auxiliar en su “cuello”. “—donde la cápside se une a la cola del virus.
En imágenes detalladas de microscopía electrónica tomadas por Tagide deCarvalho, subdirector de las Instalaciones Centrales de la Facultad de Ciencias Naturales y Matemáticas y primer autor del nuevo artículo, el 80 por ciento (40 de 50) de los ayudantes tenían un satélite atado al cuello. Algunos de los que no tenían restos de zarcillos satélite presentes en el cuello. Erill, autor principal del artículo, los describe como “marcas de mordiscos”.
“Cuando lo vi, pensé: No puedo creerlo”, dice deCarvalho. “Nadie ha visto nunca un bacteriófago, ni ningún otro virus, adherido a otro virus”.
Una relación de virus a largo plazo
Después de las observaciones iniciales, Elia Mascolo, estudiante de posgrado del grupo de investigación de Erill y coautora del artículo, analizó los genomas del satélite, el ayudante y el anfitrión, lo que reveló más pistas sobre este virus nunca antes visto. relación. La mayoría de los virus satélite contienen un gen que les permite integrarse en el material genético de la célula huésped después de ingresar a la célula. Esto permite que el satélite se reproduzca cada vez que un ayudante ingrese a la celda a partir de ese momento. La célula huésped también copia el ADN del satélite junto con el suyo cuando se divide.
Una muestra de bacteriófago de WashU también contenía un ayudante y un satélite. El satélite WashU tiene un gen para la integración y no se conecta directamente a su ayudante, de forma similar a los sistemas de satélites auxiliares observados anteriormente.
Sin embargo, el satélite de la muestra de la UMBC, denominado MiniFlayer por los estudiantes que lo aislaron, es el primer caso conocido de un satélite sin gen de integración. Debido a que no puede integrarse en el ADN de la célula huésped, debe estar cerca de su ayudante, llamado MindFlayer, cada vez que ingresa a una célula huésped si quiere sobrevivir. Dado que, aunque el equipo no demostró directamente esta explicación, “adjuntar ahora tenía mucho sentido”, dice Erill, “porque de lo contrario, ¿cómo vas a garantizar que vas a entrar en la celda al mismo tiempo?”
Análisis bioinformático adicional realizado por Mascolo y Julia López-Pérez, otra Ph.D. estudiante que trabaja con Erill, reveló que MindFlayer y MiniFlayer han estado coevolucionando durante mucho tiempo. “Este satélite ha estado sintonizando y optimizando su genoma para asociarlo con el ayudante durante, diría yo, al menos 100 millones de años”, dice Erill, lo que sugiere que puede haber muchos más casos de este tipo de relación esperando ser descubiertos. .
¿Contaminación o descubrimiento?
Este descubrimiento innovador fácilmente podría haberse pasado por alto. El proyecto comenzó como un semestre típico en el programa SEA-PHAGES, un plan de estudios de investigación en el que los estudiantes universitarios aíslan bacteriófagos de muestras ambientales, los envían para secuenciarlos y luego utilizan herramientas bioinformáticas para analizar los resultados. Cuando el laboratorio de secuenciación de la Universidad de Pittsburgh informó de contaminación en la muestra de la UMBC que se esperaba que contuviera el fago MindFlayer, el viaje comenzó.
La muestra incluía una secuencia grande: el fago que esperaban. “Pero en lugar de simplemente encontrar eso, también encontramos una pequeña secuencia, que no se correspondía con nada de lo que sabíamos”, dice Erill, quien también es uno de los líderes del programa SEA-PHAGES de la UMBC, llamado Phage Hunters, junto con Steven Caruso, profesor titular de ciencias biológicas. Caruso volvió a realizar el aislamiento, lo envió para secuenciarlo y obtuvo resultados idénticos.
Fue entonces cuando el equipo llamó a deCarvalho para obtener una imagen de lo que estaba sucediendo con el microscopio electrónico de transmisión (TEM) en Keith R. Porter Imaging Facility (KPIF) de UMBC. Sin las imágenes, el descubrimiento hubiera sido imposible.
“No todo el mundo tiene un TEM a su disposición”, señala deCarvalho. Pero con los instrumentos del KPIF, dice deCarvalho, “puedo dar seguimiento a algunas de estas observaciones y validarlas con imágenes. Hay elementos de descubrimiento que sólo podemos hacer usando el TEM”.
El descubrimiento del equipo sienta las bases para futuros trabajos para descubrir cómo se adhiere el satélite, qué tan común es este fenómeno y mucho más. “Es posible que muchos de los bacteriófagos que la gente pensaba que estaban contaminados fueran en realidad estos sistemas auxiliares de satélites”, dice deCarvalho, “así que ahora, con este artículo, la gente podría reconocer más de estos sistemas”.
Fuente: phys.org