Margarita I. Bernal Uruchurtu
Centro de Investigaciones Químicas de la UAEM. Miembro de la Academia de Ciencias de Morelos
El pasado cuatro de octubre se anunció que el Premio Nobel de Química se otorgaría este año a tres científicos “por el desarrollo de la microscopía crio-electrónica para la determinación de alta resolución de la estructura de biomoléculas en solución”. Los premiados Jacques Dubochet, Joachim Frank y Richard Henderson lograron el sueño de muchos: conseguir una imagen con detalle molecular de la estructura de una molécula en acción y no de cualquier molécula, de moléculas de gran tamaño como son algunas proteínas.
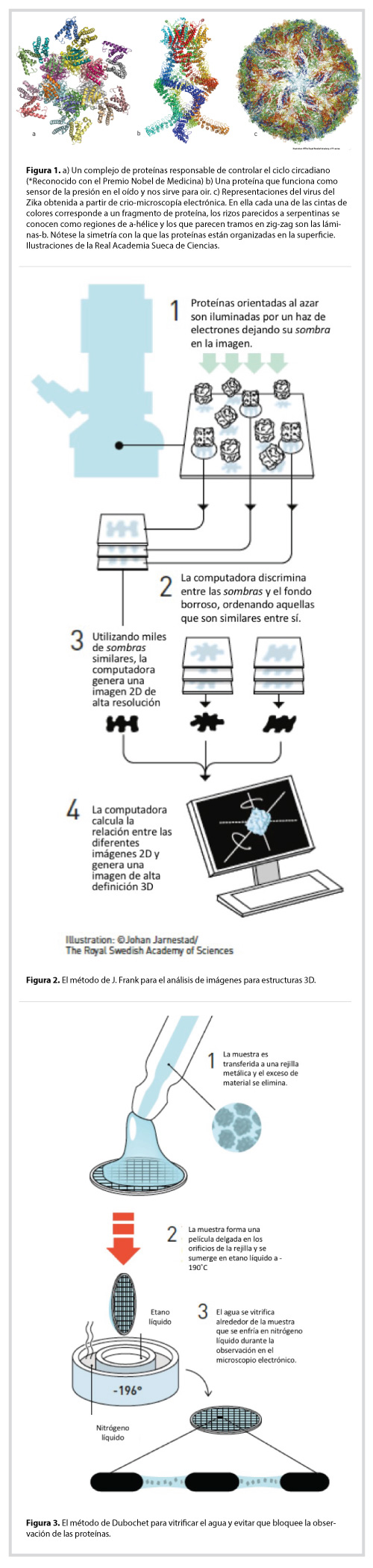
Cuando algunos investigadores comenzaron a sospechar que el virus del Zika era el responsable de la epidemia de recién nacidos con daño cerebral, utilizaron la técnica de crio-microscopía electrónica (crio-ME) para observar el virus. Después de pocos meses de trabajo lograron imágenes tridimensionales (3D) de alta resolución que son de crucial importancia para la búsqueda de sitios susceptibles de ser atacados por fármacos. En la Figura 1 se muestra una de las representaciones del virus. Este progreso es el resultado de la contribución de los equipos de trabajo de los premiados este año. Jacques Dubochet desarrolló los métodos que sirven para preparar a las muestras para los estudios de crio-ME; Joachim Frank desarrolló los métodos computacionales necesarios para el procesamiento de las imágenes de biomoléculas en solución y Richard Henderson demostró que es posible lograr imágenes con resolución atómica con esta técnica.
En ocasiones, para valorar justamente la importancia de una aportación es conveniente comparar con las alternativas para lograr resultados similares.
¿Cómo sabemos la forma de las moléculas?
 Ya en el siglo XVII se jugueteaba con la idea de que la forma de los cristales reflejaban las formas o los arreglos de las partículas que los constituían. Abbé René Just Haüy, reconocido como uno de los primeros cristalógrafos, estaba convencido que la forma de cada cristal podía relacionarse con la forma en la que se agrupaban sus partículas, a lo que el llamaba una molécula componente. Esta era una idea que venía quizá desde el siglo anterior en el que Johannes Kepler especulaba ya acerca de la simetría hexagonal de los cristales de hielo. Los químicos a principio del siglo XIX no tenían una idea clara de lo que eran las moléculas pero, los modelos atómicos más simples les eran útiles para proponer la idea de compuestos, moléculas formadas por átomos diferentes en proporciones constantes. En 1818, un químico francés Michel E. Chevreul agregaría a la distinción entre compuestos diferentes la idea de que las naturaleza de un compuesto químico dependía, además de su composición, del arreglo de los átomos en la molécula. Sin embargo, sus ideas no tuvieron mucho eco en la comunidad científica ya que no conocían ninguna técnica que les permitiese investigarlo. Aún cincuenta años después, el uso de modelos moleculares en los que los átomos se representan con esferas de madera pintadas de distintos colores para distinguir el tipo de átomo y se unen por segmentos de palos de madera, se consideraba un recurso didáctico ridículo. – Nadie puede conocer cómo están organizados los átomos.
Ya en el siglo XVII se jugueteaba con la idea de que la forma de los cristales reflejaban las formas o los arreglos de las partículas que los constituían. Abbé René Just Haüy, reconocido como uno de los primeros cristalógrafos, estaba convencido que la forma de cada cristal podía relacionarse con la forma en la que se agrupaban sus partículas, a lo que el llamaba una molécula componente. Esta era una idea que venía quizá desde el siglo anterior en el que Johannes Kepler especulaba ya acerca de la simetría hexagonal de los cristales de hielo. Los químicos a principio del siglo XIX no tenían una idea clara de lo que eran las moléculas pero, los modelos atómicos más simples les eran útiles para proponer la idea de compuestos, moléculas formadas por átomos diferentes en proporciones constantes. En 1818, un químico francés Michel E. Chevreul agregaría a la distinción entre compuestos diferentes la idea de que las naturaleza de un compuesto químico dependía, además de su composición, del arreglo de los átomos en la molécula. Sin embargo, sus ideas no tuvieron mucho eco en la comunidad científica ya que no conocían ninguna técnica que les permitiese investigarlo. Aún cincuenta años después, el uso de modelos moleculares en los que los átomos se representan con esferas de madera pintadas de distintos colores para distinguir el tipo de átomo y se unen por segmentos de palos de madera, se consideraba un recurso didáctico ridículo. – Nadie puede conocer cómo están organizados los átomos.
En la primera mitad del siglo XX los científicos sabían que moléculas como las proteínas, el ADN y el ARN eran fundamentales para el funcionamiento de la célula pero no tenían ninguna idea sobre su forma. Aunque en ese momento era ya posible tener una buena idea de la estructura de muchos compuestos químicos, el tamaño de las moléculas biológicas, macromoléculas, dificultaba enormemente la aplicación de las técnicas exitosas para la determinación de la estructura de compuestos inorgánicos u orgánicos.
Los estudiantes de los primeros años de las licenciaturas en el área de química aprenden que existen numerosas técnicas para el estudio de: la composición de un compuesto químico, la organización de los átomos en grupos químicos dentro de las moléculas y la estructura tridimensional de las moléculas. Cada una de estas técnicas es, en términos coloquiales, una forma de interrogatorio a la naturaleza. Las preguntas se formulan empleando radiación electromagnética y las respuestas son interpretadas dependiendo del efecto que la radiación tiene sobre las moléculas. Recordemos que la radiación electromagnética podemos clasificarla según el tamaño de la onda o la cantidad de energía que transporta. La energía de la radiación infrarroja con longitudes de onda entre 2.5 -25 mm (micrómetro=milésimas de milímetro) provoca la vibración de los enlaces entre los átomos, así podemos conocer cómo están unidos o agrupados los átomos y aunque es muy útil, no nos da una imagen de la estructura de la molécula. La luz visible, con ondas mil veces más pequeñas que la infrarroja, entre 400 – 800 nm (nanómetros= millonésimas de milímetro) y la ultra violeta (150-400 nm) es de mayor energía y por ello modifica el estado en el que se encuentran los electrones en los átomos. Un electrón absorbe un fotón de la radiación y pasa a otro estado de mayor energía, al regresar al estado original desprende un fotón cuya energía corresponde a la diferencia entre los dos estados. Así se puede conocer qué tipo de átomos y enlaces químicos tiene una molécula pero, nuevamente, no nos da información sobre la estructura. Los rayos X son radiación con menor longitud de onda que los anteriores, entre 0.01 a 10 nm, esta longitud es de un tamaño comparable con la distancia entre los átomos de una molécula por ello, cuando un cristal es irradiado con rayos X se produce un fenómeno conocido como difracción. Este fenómeno ocurre al incidir la onda sobre cada uno de los átomos que forman el cristal y difundir estos la onda. La interferencia entre la onda inicial y las que se difunden produce un patrón que puede ser analizado para conocer la posición de los átomos. La técnica que utiliza este principio físico, la difracción de rayos X, si permite conocer la estructura de las moléculas en particular cuando estas estén ordenadas en un arreglo cristalino. Además, de ofrecer una estructura global de la molécula, nos proporciona valores exactos de las longitudes y ángulos de enlace, detalles de conformación, configuración absoluta de la molécula, empacado molecular e interacciones intermoleculares.
Además de las técnicas que sirven para indagar la naturaleza química de las moléculas, la microscopía avanzó durante el siglo XX para lograr imágenes de objetos que nuestra visión no alcanza. Por supuesto que la biología fue enormemente transformada con la posibilidad de tener imágenes de tejidos, organismos unicelulares, células y sus componentes. Una gran cantidad de técnicas se desarrollaron para mejorar la calidad de las imágenes obtenidas con microscopios cada vez con mejor resolución. La resolución de las imágenes es la posibilidad de distinguir dos objetos cercanos como unidades independientes y depende directamente de la longitud de onda utilizada para iluminar el objeto. Aumentar la resolución es posible hasta el límite en el que el fenómeno de difracción aparece. Los límites tradicionales de la resolución óptica en microscopía son cercanos a los 0.2 micrómetros. Obtener imágenes cada vez con mayor resolución requiere la utilización de una fuente de iluminación con menor longitud de onda. Así, en la década de 1930 se desarrolló la posibilidad de iluminar la muestra con un rayo o haz de electrones cuya longitud de onda es muy pequeña. Este tipo de iluminación atraviesa la muestra y, del otro lado de ella, se detectan los electrones que han sido desviados de su trayectoria original por el material y cuya nueva trayectoria contiene información útil para generar una imagen de la muestra. Al ser tan pequeña su longitud de onda, pueden iluminar objetos que la luz visible no puede pero, esa longitud de onda los hace tener una gran cantidad de energía que puede destruir a su paso moléculas delicadas y tejidos. Es una técnica intrínsecamente dañina. El reto: ¿Cómo obtener suficiente información antes de dañar la molécula que observas?
Los retos de estudiar macromoléculas
El fuerte desarrollo de la técnica de difracción de rayos X, indiscutible estrella en la determinación de estructuras moleculares, a partir de la década de 1950 es motivado por la frustración con la técnica de microscopía electrónica. En esta última, las muestras deben encontrarse al vacío (para evitar que las moléculas del aire interfieran con el haz de electrones). Al hacer esto, las moléculas biológicas pierden su estructura original ya que el agua en la que están disueltas sale de la estructura y ésta se colapsa. Además del daño que describimos en la sección anterior. Lograr cristalizar una proteína fue uno de los principales retos. Las macromoléculas existen, funcionan y son estables en medios acuosos, sacarlas de su medio natural afecta su estructura. Al “obligar” a las proteínas a formar un cristal, lo que se busca es que muchas copias de la misma molécula se empaquen en una conformación estable y fija. En este proceso, aunque se pierde el agua que las rodea, el pequeño cristal que se forma puede ser iluminado con rayos X para generar el patrón de difracción que conduce a su estructura molecular.
En la década de 1960 se estableció la estructura de las primeras proteínas. En 1962 Max Perutz y John Kendrew reciben el Premio Nobel, por sus estudios de proteínas globulares como la mioglobina y la hemoglobina. En 1964 Dorothy Crowfoot Hodgkin recibió el Premio Nobel por la determinación de la estructura de importantes substancias bioquímicas.
En la década de 1980, el uso de la resonancia magnética nuclear (RMN) se suma al reto no solo de conocer la estructura de proteínas en estado sólido sino, también en disolución. Esta técnica, además de revelar su estructura, también nos da una importante información acerca del movimiento y la interacción con otras moléculas. Su limitación: su uso está limitado en disolución a proteínas relativamente pequeñas. Gracias al uso combinado de la difracción de rayos X y RMN, actualmente existen bases de datos con información sobre miles de modelos de biomoléculas que se utilizan intensivamente para la investigación básica y el desarrollo farmacéutico.
En el Centro de Investigaciones Químicas (CIQ) de la UAEM se encuentra el Laboratorio Nacional de Estructura de Macromoléculas (LANEM). Este proyecto conjunto con el Instituto de Química de la UNAM permitió desde hace casi una década contar con el equipamiento necesario para estudiar las macromoléculas: un equipo de difracción de rayos X y uno de RMN de 700 MHz (el más grande en México); además de los equipos de todas las técnicas complementarias. El financiamiento necesario para la instalación; operación y mantenimiento de esta infraestructura ha sido logrado con las contribuciones del CONACYT, la UNAM y la UAEM. (A pesar de que el equipo de RMN se dañó durante el sismo del pasado 19 de septiembre, el CIQ está haciendo las gestiones necesarias para lograr reponer este equipo en el corto plazo).
Pero el reto allí sigue: proteínas grandes y complejos con varias unidades, por ejemplo los que se encuentran insertados en la membrana celular, no cristalizan y sus tallas están más allá de la capacidad de la técnica de NMR. ¿Cómo conocer entonces su estructura?
La crio-microscopía electrónica
Uno de los ganadores del Premio Nobel de este año, Richard Henderson, inició su carrera científica como cristalógrafo de Rayos X de proteínas. Su frustración al no lograr cristalizar una proteína de membrana lo condujo a probar otra técnica, la microscopía electrónica. Por supuesto que muchos colegas le dijeron que eso no era una buena idea. Los electrones incineraría su proteína rápidamente y si disminuía la energía de los electrones, sólo lograría una imagen borrosa.
La visión de Henderson consistió en la elección de su proteína: la bacteriorodopsina. Una proteína que se encuentra en la membrana de organismos fotosintéticos y que captura la radiación solar. Al colocar un pedazo de membrana que contenía la proteína bajo el microscopio electrónico, esta no se descompuso; claro que el modelo que de ella se obtuvo en 1975 era bastante burdo pero era un rayo de esperanza. La imagen de la bacteriorodopsina se construyó tomando imágenes a diferentes ángulos con lo que se observó que la proteína subía y bajaba a través de la membrana siete veces; parecían siete pilares deformados y era, en ese momento, la mejor imagen de una proteína jamás obtenida, alcanzó una resolución de 0.7 nm. El resultado era bueno, pero los cristalógrafos de rayos X de la época alcanzaban ya resoluciones de 0.3 nm ¿Sería posible mejorar la técnica? ¿Sería posible utilizarla para proteínas que no estuviesen rigidizadas por la membrana celular? Pocos creían en la posibilidad de hacerlo.
Ese mismo año, Joachim Frank propuso teóricamente una técnica que podría, a partir de la superposición de imágenes 2D, reconstruir la estructura 3D de un objeto con gran resolución. Diez años después lo consiguió. Su estrategia está basada en un método matemático que permite a una computadora distinguir patrones similares obtenidos con imágenes borrosas provenientes de un microscopio electrónico. En la Figura 2 se muestra una versión simplificada de su técnica. Su primera publicación del uso de esta técnica mostraba un modelo de la superficie de un ribosoma, la fábrica de proteínas de las células. En 1990, el grupo de Henderson mostró que era posible mejorar la resolución de la imagen al promediar sobre muchas copias del mismo objeto. Su trabajo sobre la bacteriorodopsina alcanzaba a bajas temperaturas una mucho mejor resolución.
Por su parte, Jacques Dubochet estaba, en esta misma época, ocupado con el problema de cómo rigidizar una proteína en solución. A Henderson le ayudó que la bacteriorodopsina estuviese contenida por la membrana y evitó que se secara poniéndola en una solución de glucosa que evitaba la rápida evaporación del agua al colocarla en vacío. La idea de Dubochet fue enfriar el agua. Sin embargo, la formación de hielo era en sí otro problema ya que los pequeñísimos cristales de hielo difractan los electrones y al hacerlo borran las señales provenientes de la proteína. Varios científicos trabajaron desde treinta años antes en descubrir cómo enfriar las muestras biológicas sin que se formaran cristales de hielo. Para lograr esto es indispensable enfriar muy rápidamente de forma que las moléculas permanezcan en la posición en la que se encuentran en la disolución. Al hacer crio-microscopía electrónica se esperaba otra gran ventaja: disminuir el daño que los electrones hacen en la muestra observada.
A principios de la década de 1980, se pensaba que la rapidez necesaria para enfriar el agua y evitar la formación de cristales no era alcanzable. Sin embargo en 1981 Dubochet mostró la forma en la que se podía formar una película de agua vitrificada, es decir, en estado no cristalino. El agua se deposita sobre una película de carbón utilizando un atomizador y rápidamente se sumerge en etano o propano líquido no usualmente a -190˚C y se mantiene a baja temperatura utilizando nitrógeno líquido; así se lograba una transmisión de electrones prácticamente uniforme. Además, al conservar la muestra a temperaturas inferiores a -160˚C la muestra podía observarse por periodos más prolongados sin descomposición. En la Figura 3 se muestra un esquema de la técnica de vitrificación de agua. Este método resolvió el problema que desde 1950 impedía el uso del microscopio electrónico para el estudio de sistemas vivos.
Evidentemente, muchos otros avances científicos y tecnológicos concurren a mejorar las imágenes de las macromoléculas: mejores detectores de los electrones; computadoras más rápidas para el análisis de imágenes.
La posibilidad de estudiar proteínas sin necesidad de cristalizarlas nos ofrece el retrato detallado de una molécula en acción. La crio-ME revela la estructura de moléculas cada vez más grandes por lo que las grandes agencias de investigación comienzan ya a invertir en centros dedicados a esta técnica con lo que quizá, instalaciones enormes como los sincrotrones en donde se cuenta con haces intensos de rayos X para estudios de difracción de cristales caerán, tendrán mucho menos demanda.
Para conocer más:
- https://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/popular-chemistryprize2017.pdf
- Video: Serious Science (20 June 2017) Electron Cryomicroscopy — Richard Henderson: http: www.youtube.com/watch?v=L6U–sYUF9s
Fuentes consultadas:
- C. Bertozzi, Atoms out of Blobs: CryoEM takes the Nobel Prize in Chemistry, ACS Central Science, 3, 1056 (2017)
- E. Gebel Berg, Breaking the Cristal Ceiling, ACS Central Science, 1, 283-285, (2015)
- R. F. Service y E. Stokstad, Cold clear view of molecules nets chemistry prize, Science, 358, 156-157 (2017)
- «The Nobel Prize in Physics 2017 – Scientific Background: The development of cryo-electron microscopy»Nobelprize.org. Nobel Media AB 2014. Web. 27 Oct 2017. <http://www.nobelprize.org/nobel_prizes/chemistry/laureates/2017/advanced.html>
Fuente: Academia de Ciencias de Morelos

