Marco Antonio Rogel, Mónica Rosenblueth, Julio Martínez, Jacob Banuelos, Esperanza Martínez Romero
En ingeniería genética se obtienen y producen genes (unidades de información genética) de interés biotecnológico o científico, mediante un proceso al que se le denominó clonación.
“entonces clonar pa(ra) qué?”
Eso escribió el ahora famoso director de teatro Alberto Lomnitz en un cuaderno de un laboratorio en el Instituto de Investigaciones Biomédicas de la UNAM hace muchos años. Esta pregunta marcaba el final de un muy pequeño texto. Entonces era el principio de la era del ADN recombinante. Julio Martínez (padre de una de las coautoras de este artículo) respondió a esa pregunta años más tarde, cuando se maravillaba que podía inyectar insulina humana a su esposa que tenía diabetes. La insulina era producida en bacterias gracias a que el gen de humano para la producción de insulina había sido clonado en bacterias, mediante metodología de ADN recombinante (ver artículo del Dr. Agustín López-Munguía en este número de la revista). La utilidad de esta tecnología en medicina es muy reconocida, pero es menos conocido su impacto en el desarrollo de la ciencia, en especial en la microbiología, en donde ha permitido reconocer una amplísima diversidad de microbios.
Los animales estamos colonizados de por vida y de manera natural por microbios que en su mayoría no pueden ser cultivados en los laboratorios, por lo que, durante mucho tiempo, fueron desconocidos. Gracias a las secuencias nucleotídicas (el orden de los núclétidos Adenina, Timina, Guanina y Citosina, que constituyen el ADN) de los genes clonados a partir del ADN extraído directamente de los hospederos (animales o plantas) o del ambiente, podemos conocer de la existencia microbiana sin necesidad de cultivos en laboratorio. Así surgió la metagenómica, que es una rama muy próspera de las Ciencias Genómicas [1]. De allí aprendimos que los microbios pueden proteger a los animales o plantas contra patógenos o condiciones adversas. Además, algunas bacterias de plantas y también de insectos pueden proveer los aminoácidos y vitaminas que no producen los hospederos y favorecer así su crecimiento y reproducción.
El éxito evolutivo de los insectos se atribuye en parte a sus microbios asociados. La lista de bacterias encontradas en insectos e identificados con enfoques de ADN recombinante es larga [2] y llama la atención que las bacterias están en general especializadas sólo a algún tipo de insecto. Las termitas que se alimentan de madera pueden crecer gracias a que en su intestino tienen microbios que degradan la madera y bacterias que fijan nitrógeno, permitiéndoles compensar los niveles pobres de nitrógeno en la madera. La identidad de los fijadores de nitrógeno se reveló con técnicas de ADN recombinante [3].
En el Centro de Ciencias Genómicas de la UNAM, con un enfoque independiente de cultivo y usando ADN recombinante, descubrimos una nueva bacteria capaz de fijar nitrógeno dentro de las cochinillas del carmín (Figura 1) [4; 5] que se usan desde tiempos prehispánicos (ver BiotecMov No. 9 ) para producir un pigmento rojo utilizado en la industria. La fijación de nitrógeno por bacterias dentro de la cochinilla permite a los insectos vivir y reproducirse en las pencas de nopal las cuales son muy pobres en nitrógeno.
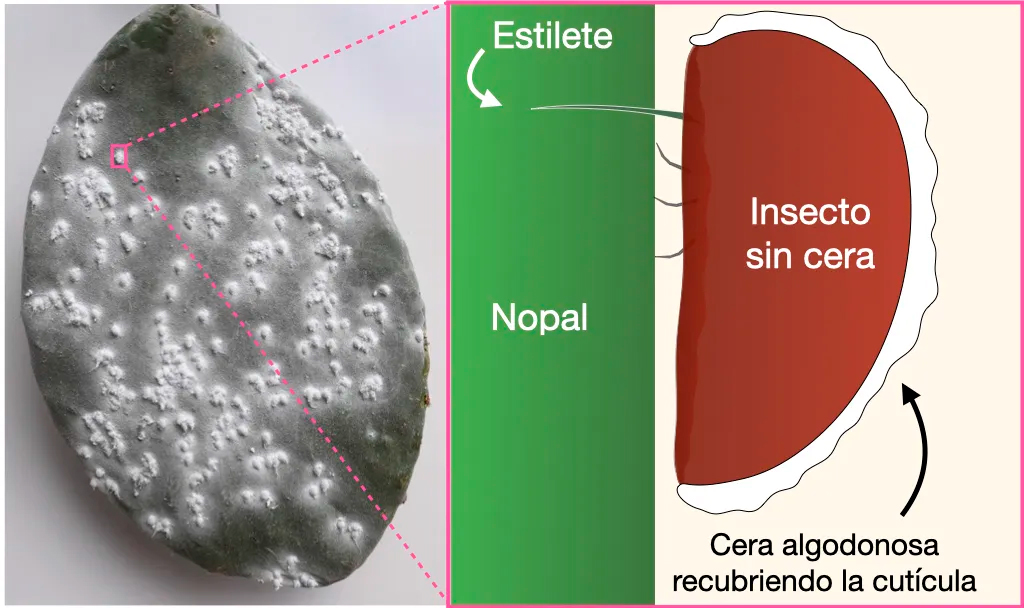
La identidad certera de algunos simbiontes de plantas (como Rhizobium), los cuáles sí pueden ser cultivados en laboratorio, se logró mediante la síntesis de genes por PCR (Reacción en Cadena de la Polimerasa, por sus siglas en inglés, muy conocida ahora desde la pandemia), seguida de clonación y secuenciación (detalles de las técnicas de secuenciación pueden ser consultadas en BiotecMov No. 13 ). Así pudimos describir varias especies nuevas de bacterias que forman nódulos en las raíces de plantas leguminosas que son nativas de México. Los efectos benéficos de los rizobios sobre el crecimiento de las plantas se han constatado en los campos agrícolas y en laboratorio (Figura 2).

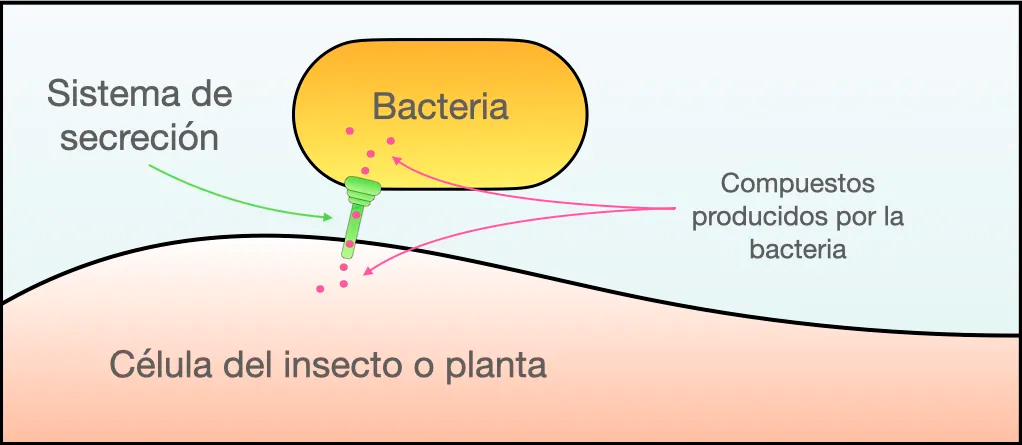
Por otro lado, con el fin de descubrir las funciones de los microbios dentro de animales y plantas se requiere estudiar mutantes generadas a partir de los genes clonados. Estas metodologías han permitido identificar muchos tipos de genes que dictan funciones muy diversas. Aquí destacamos a los sistemas de secreción [6] que se encuentran tanto en bacterias simbiontes de plantas, como en las de insectos, y pueden suprimir las respuestas de defensa de las plantas y ser determinantes de la especificidad por planta (esto es, que bacterias específicas sólo estén presentes en ciertas plantas) [7]. La bacteria Sodalis es benéfica para algunos insectos y les puede proporcionar amino acidos esenciales. Para la colonización de uno de estos insectos, Sodalis requiere de los sistemas de secreción [8,9]. Los sistemas de secreción son fascinantes por ser minúsculas jeringas mediante las cuales las bacterias inyectan diversas moléculas a animales o a plantas (Figura 3). También las bacterias patógenas que causan enfermedades en plantas o animales se valen de los sistemas de secreción y utilizan las mismas jeringas pero para inyectar tóxinas y otras sustancias nocivas. De hecho, los sistemas de secreción se descubrieron en bacterias patógenas donde son ampliamente estudiados.
Al igual que lo que ocurre con algunas bacterias, los hongos de la micorriza, que constituyen raíces adicionales en las plantas, no pueden ser cultivados en medios de laboratorio y su identificación se ha logrado con técnicas de ADN recombinante. La micorriza se considera la madre de todas las simbiosis de plantas y se presume que estos hongos han sido simbiontes de plantas por cerca de 400 millones de años.
Es claro que el enfoque de ADN recombinante ha revelado una enorme diversidad microbiana y nuevas especies de microbios asociadas a plantas y animales y algunas de sus funciones. Sin embargo, todavía nos falta aprender más para poder entender y utilizar óptimamente a los microbios en beneficio de plantas y animales.
Referencias
- Handelsman, J., Rondon, M. R., Brady, S. F., Clardy, J., & Goodman, R. M. (1998). Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products. Chemistry & biology, 5\ , R245-R249.
- Moran, N., & Baumann, P. (1994). Phylogenetics of cytoplasmically inherited microorganisms of arthropods. Trends in Ecology & Evolution, 9\ , 15-20.
- Ohkuma, M., Noda, S., & Kudo, T. (1999). Phylogenetic diversity of nitrogen fixation genes in the symbiotic microbial community in the gut of diverse termites. Applied and Environmental Microbiology, 65\ , 4926-4934.
- Ramírez-Puebla, S. T., Rosenblueth, M., Chávez-Moreno, C. K., Catanho Pereira de Lyra, M. C., Tecante, A., & Martínez-Romero, E. (2010). Molecular phylogeny of the genus Dactylopius (Hemiptera: Dactylopiidae) and identification of the symbiotic bacteria. Environmental Entomology, 39\ , 1178-1183.
- Vera-Ponce de León, A., Ormeno-Orrillo, E., Ramírez-Puebla, S. T., Rosenblueth, M., Degli Esposti, M., Martínez-Romero, J., & Martínez-Romero, E. (2017). Candidatus Dactylopiibacterium carminicum, a nitrogen-fixing symbiont of Dactylopius cochineal insects (Hemiptera: Coccoidea: Dactylopiidae). Genome Biology and Evolution, 9\ , 2237-2250.
- Teulet, A., Camuel, A., Perret, X., & Giraud, E. (2022). The versatile roles of type III secretion systems in rhizobium-legume symbioses. Annual Review of Microbiology, 76\ , 45-65.
- Hernández-Oaxaca, D., Claro, K., Rogel, M. A., Rosenblueth, M., Martinez-Romero, J., & Martinez-Romero, E. (2023). Novel symbiovars ingae, lysilomae and lysilomaefficiens in bradyrhizobia from tree-legume nodules. Systematic and Applied Microbioly, 46\ , 126433.
- Dale, C., Young, S. A., Haydon, D. T., & Welburn, S. C. (2001). The insect endosymbiont Sodalis glossinidius utilizes a type III secretion system for cell invasion. Proceedings of the National Academy of Sciences, 98\ , 1883-1888.
- Rosas-Pérez, T., Vera-Ponce de León, A., Rosenblueth, M., Ramírez-Puebla, S. T., Rincón-Rosales, R., Martínez-Romero, J., Dunn, M. F., Kondorosi, E. & Martínez-Romero, É. (2017). The symbiome of Llaveia cochineals (Hemiptera: Coccoidea: Monophlebidae) includes a gammaproteobacterial cosymbiont Sodalis TME1 and the known Candidatus Walczuchella monophlebidarum. En: Insect Physiology and Ecology*, Vonnie D.C. Shield (Ed.) pp 115-134.
Fuente: biotecmov.ibt.unam.mx

