El ADN contiene la información genética que dirige el desarrollo de los organismos y regula las funciones vitales. La longitud de la molécula de ADN supera en muchos órdenes de magnitud las dimensiones del núcleo celular, por lo que debe compactarse enormemente y de forma muy precisa para poder acomodarse en su interior.
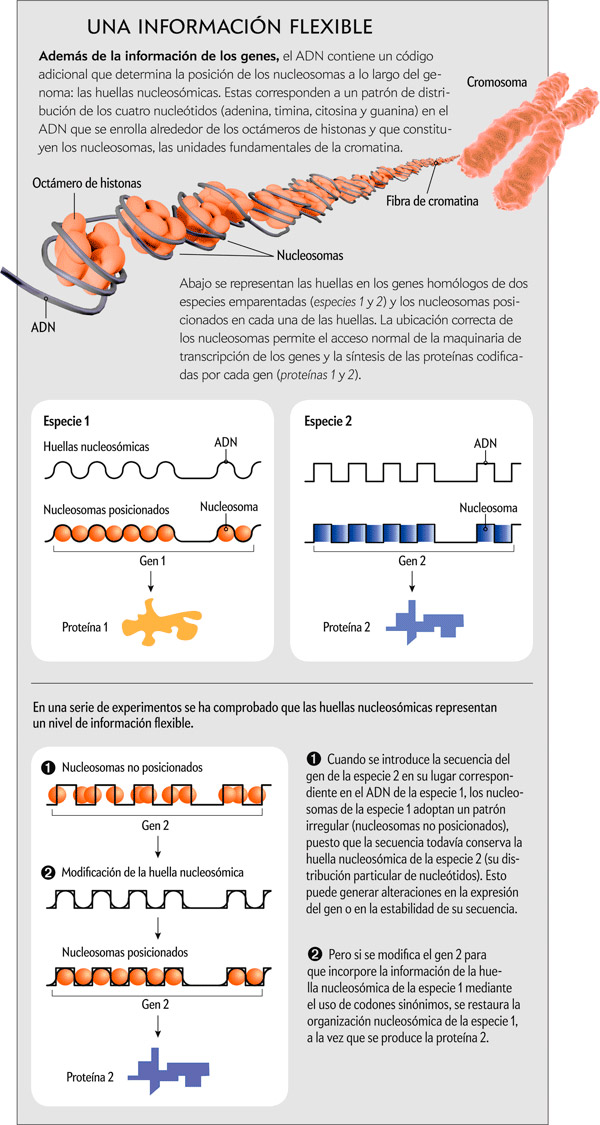
Este empaquetamiento resulta posible gracias a la asociación del ADN con ciertas proteínas estructurales, llamadas histonas, en la fibra de cromatina. Las unidades fundamentales de la cromatina son los nucleosomas, complejos de ocho histonas sobre los que se enrolla un fragmento de ADN de cadena doble. Dicho fragmento está formado por 147 nucleótidos y se denomina ADN mononucleosómico. La mayoría de los nucleosomas ocupa posiciones muy estables a lo largo del genoma. Esta ubicación precisa resulta esencial porque determina la accesibilidad al ADN de las proteínas que regulan su transcripción y otras funciones.
Se sabía que la estabilidad y la dinámica de los nucleosomas dependen, en parte, de los complejos remodeladores de cromatina, un conjunto de proteínas que tiene la capacidad de reposicionar los nucleosomas. Se ha comprobado asimismo que los factores de transcripción participan también en la organización de la cromatina.
En comparación con el papel bien establecido de los remodeladores y de los factores de transcripción, la contribución de la secuencia del ADN al posicionamiento de los nucleosomas resulta más controvertida. Varios grupos de investigación han descrito que la distribución de algunos dinucleótidos (dos nucleótidos consecutivos en una de las dos hebras del ADN) en el ADN de un nucleosoma, en concreto los de adenina y timina y los de citosina y guanina (los cuatro componentes fundamentales del ADN), favorece la torsión alrededor del octámero de histonas. Ninguno de esos estudios, sin embargo, ha detectado elementos capaces de conducir los nucleosomas hasta las regiones que ocupan a lo largo del genoma. En nuestro laboratorio nos propusimos descifrar ese código de información adicional del ADN que determina la posición de los nucleosomas, así como averiguar el modo en que esa información influye en la expresión de los genes.
Identificación de las huellas
Aprovechando que las levaduras son los organismos en los que se ha determinado con mayor resolución la distribución de los nucleosomas en el genoma, iniciamos la búsqueda de las señales que dirigen su ubicación en la levadura Schizosaccharomyces pombe. Analizamos unas 40.000 secuencias de ADN asociadas a nucleosomas que ocupaban posiciones muy estables en el genoma. A continuación, calculamos la proporción de cada uno de los cuatro nucleótidos en las 147 posiciones del ADN mononucleosómico y obtuvimos una distribución muy bien definida y diferente para cada nucleótido. Por otro lado, el análisis del mismo número de secuencias de S. pombe de la misma longitud pero seleccionadas al azar generó distribuciones homogéneas coincidentes con la frecuencia de cada nucleótido en el genoma. Ello demostraba que la distribución singular hallada de los nucleótidos aparecía solo en las secuencias del ADN enrollado sobre los octámeros de histonas. Denominamos huellas nucleosómicas (nucleosomal signatures) al patrón promedio de distribución de los cuatro nucleótidos a lo largo del ADN que está asociado a un nucleosoma.
Para investigar si tales huellas estaban presentes también en otros genomas, realizamos el mismo análisis en varias especies de levaduras y observamos que, además de existir, adoptaban una forma diferente en cada una de ellas, a pesar de la gran conservación de las histonas en las distintas especies.
Con estas observaciones nos preguntamos cuál era el origen de estas señales y si desempeñaban alguna función biológica. En particular, nos interesaba saber si la información contenida en ellas contribuiría a que los nucleosomas ocupasen posiciones específicas en el genoma. Para resolver esa incógnita era necesario distinguir si las huellas nucleosómicas existían como consecuencia del posicionamiento de los nucleosomas en el genoma o eran ellas las que causaban o dirigían ese posicionamiento.
Cada una de esas dos hipótesis hacía predicciones diferentes respecto a la relación de causalidad entre las huellas nucleosómicas y la localización de los nucleosomas, que analizamos experimentalmente en nuestro laboratorio. En el primer caso, si la información del ADN no contribuyese a la organización de los nucleosomas, esta última no variaría al modificar las secuencias asociadas a ellos. En el segundo caso, si la secuencia contribuyera al posicionamiento, existiría la posibilidad de diseñar, a partir de la información conocida de las huellas nucleosómicas, secuencias de ADN artificiales con capacidad para dirigir los nucleosomas a posiciones específicas del genoma.
Funcionamiento de las huellas
Para analizar la primera hipótesis, sintetizamos varios fragmentos de ADN mononucleosómico en los que cambiamos el orden de los nucleótidos respecto a las secuencias nativas y, a continuación, reemplazamos tales secuencias por los fragmentos artificiales. A continuación, se digirió la cromatina con una enzima (nucleasa micrococal) que corta las regiones entre los nucleosomas y permite identificar el posicionamiento de estos en genes específicos o en el genoma entero. El resultado fue una alteración de la distribución de los nucleosomas que coincidía exactamente con las zonas modificadas y que tenía lugar tanto en regiones transcritas (codificantes y no codificantes) como no transcritas. Ello sugería que habíamos eliminado algún tipo de información necesaria para el posicionamiento y que, por lo tanto, la secuencia parecía contribuir al mismo. A pesar de ello, no podíamos concluir que esa información estuviese codificada en las huellas nucleosómicas.
Con el fin de poner a prueba la segunda hipótesis, incorporamos información derivada de las huellas nucleosómicas de S. pombe y S. cerevisiae en moléculas de ADN artificiales no codificantes y las introdujimos en sus genomas. El análisis de las cepas resultantes reveló que los nucleosomas se situaban exactamente sobre las posiciones predichas en cada especie. Sin embargo, las mismas secuencias eran incapaces de posicionar los nucleosomas cuando estas se intercambiaban entre las dos levaduras. Tales resultados demostraban que las huellas nucleosómicas contenían información para dirigir la distribución de los nucleosomas y que, además, esa información era exclusiva de cada especie, ya que no era interpretada correctamente por los nucleosomas de otras especies.
Las huellas nucleosómicas no solo están definidas por la secuencia en sí del ADN mononucleosómico, sino también por la distribución promedio de los cuatro nucleótidos a lo largo de él. Ello permite generar muchas secuencias diferentes que mantienen la capacidad para dirigir el posicionamiento de los nucleosomas.
En principio, esa flexibilidad abría la posibilidad de modificar la secuencia de los genes de una especie para que adoptasen la organización nucleosómica de los de otra, al tiempo que mantenían su capacidad de codificación original. Esto se debe a que un mismo aminoácido puede estar codificado por distintos codones sinónimos (un codón corresponde a la combinación de tres nucleótidos; la mayoría de los 20 aminoácidos existentes pueden estar codificados por 2, 3, 4 o 6 codones sinónimos). Para explorar esa posibilidad sustituimos los codones de tres genes (dos eucarióticos y uno de origen procariótico) por sus codones sinónimos y que tuvieran una composición de nucleótidos parecida a la definida por las huellas nucleosómicas de S. pombe y S. cerevisiae. Los resultados que obtuvimos al integrarlos en sus genomas revelaron que, efectivamente, los genes de un organismo, incluso los de origen procariótico, pueden modificarse para que adopten una organización nucleosómica indistinguible de los del genoma de la especie en la que se integren.
Las huellas nucleosómicas representan, pues, un nuevo nivel de información del ADN que ofrece la posibilidad de diseñar moléculas de ADN capaces de especificar la organización básica del genoma. Será interesante explorar su posible aplicación al diseño de genes y vectores de interés biotecnológico, como una herramienta para valorar la expresión y estabilidad de estos. A mayor escala, también podrían ser relevantes en el diseño de genomas sintéticos y en el campo emergente de la ingeniería de la cromatina.
Fuente: investigacionyciencia.es