Los premios Nobel de Química 2018: La ingeniería de proteínas en un tubo de ensayo
Joaquín Ramírez Ramírez
El M.C. Joaquín Ramírez Ramírez es estudiante de Doctorado del Posgrado en Ciencias Bioquímicas del Instituto de Biotecnología de la UNAM. Su trabajo doctoral trata del estudio de los factores moleculares que determinan la capacidad oxidante de una peroxigenasa fúngica. Esta publicación fue revisada por el comité editorial de la Academia de Ciencias de Morelos.
El miércoles 3 de octubre, un día después de que la Real Academia de las Ciencias de Suecia otorgara el Premio Nobel de Física (http://www.acmor.org.mx/?q=content/los-premios-nobel-de-f%C3%ADsica-2018-pinzas-de-luz-y-pulsos-luminosos-ultracortos), se anunció el galardón de Química, cuyos laureados fueron Frances H. Arnold (Estados Unidos), por un lado, y George P. Smith (Estados Unidos) y Gregory P. Winter (Reino Unido), por otro. Gracias a sus descubrimientos en el área de la genética, se ha logrado el desarrollo de proteínas que pueden resolver problemas químicos en pro de la humanidad.
 Arnold ha sido distinguida por su trabajo relacionado con la “evolución dirigida de las enzimas”, mientras que de Smith y Winter se ha reconocido su labor en la “presentación en fagos de péptidos y anticuerpos”. Una enzima es una proteína que puede catalizar reacciones químicas de una manera muy eficiente. En el caso de los fagos, son virus que pueden infectar bacterias y que han sido ampliamente utilizados para poder producir proteínas de manera artificial, las cuales pueden ser antígenos de algún patógeno y que pueden ser utilizados para fines farmacéuticos, como la producción de vacunas (http://www.acmor.org.mx/?q=content/%C2%BFvacunarse-o-no-vacunarse-esto-no-es-un-dilema).
Arnold ha sido distinguida por su trabajo relacionado con la “evolución dirigida de las enzimas”, mientras que de Smith y Winter se ha reconocido su labor en la “presentación en fagos de péptidos y anticuerpos”. Una enzima es una proteína que puede catalizar reacciones químicas de una manera muy eficiente. En el caso de los fagos, son virus que pueden infectar bacterias y que han sido ampliamente utilizados para poder producir proteínas de manera artificial, las cuales pueden ser antígenos de algún patógeno y que pueden ser utilizados para fines farmacéuticos, como la producción de vacunas (http://www.acmor.org.mx/?q=content/%C2%BFvacunarse-o-no-vacunarse-esto-no-es-un-dilema).
La evolución dirigida de las enzimas
Charles Darwin propuso en 1859 el mecanismo por el cual las especies cambian a lo largo del tiempo: la selección natural. En su época, Darwin y sus contemporáneos no sabían que las características biológicas de un ser vivo estaban determinadas por la información genética codificada en la molécula de ADN. De manera natural, esta información puede mutar o cambiar por diferentes mecanismos. Esto incrementa la diversidad de genes y la selección natural puede trabajar sobre ellos para que los genes más favorables para la supervivencia (y reproducción) de un ser vivo se propaguen en la población. Con el tiempo, los genes seleccionados serán “mejores” que los anteriores; es decir, estarán más adaptados u optimizados a las condiciones ambientales a las que están expuestos. Este proceso de evolución ha ocurrido desde que comenzó el fenómeno de la vida, hace unos 3,600 millones de años.
En las últimas décadas, el avance de las técnicas del ADN recombinante y herramientas que permiten procesar una gran cantidad de muestras, han permitido el desarrollo de la llamada evolución molecular dirigida. En particular, la evolución dirigida de proteínas o enzimas ha permitido ampliar las capacidades naturales de estas moléculas y se han mejorado con miras a una aplicación en distintos procesos industriales. Las enzimas son catalizadores de origen biológico, es decir, son moléculas que producen las células para acelerar las reacciones químicas necesarias para que ocurra el fenómeno de la vida. Las células producen muchísimas enzimas distintas y cada una de ellas tiene una estructura tridimensional que determina su función. Esta estructura o forma de la enzima está determinada a su vez por la secuencia de sus componentes elementales: los aminoácidos. En los seres vivos casi todas las proteínas están formadas por 20 distintos aminoácidos que, en diferentes combinaciones y proporciones, le confieren una identidad. Las posibles secuencias de aminoácidos tienen un universo vastísimo de posibilidades. Por ejemplo, una molécula pequeña de proteína, formada por 100 aminoácidos, puede tener 10130posibles secuencias, un número más grande que el número total de átomos en el universo.
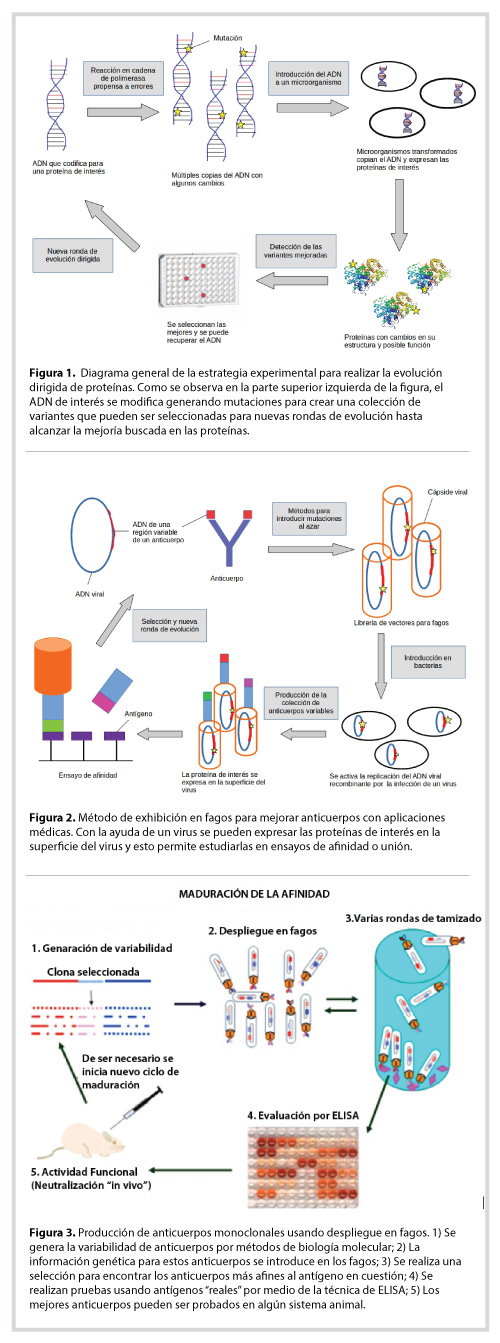
La evolución molecular dirigida consiste en replicar la evolución de moléculas como las proteínas, pero en el laboratorio, con el fin de mejorarlas, adaptarlas o simplemente entender la relación entre estructura y función, y en una escala de tiempo de días o semanas (Figura 1). El gen que codifica para una proteína o enzima en particular se puede mutar por diferentes técnicas (ahora) comunes en el laboratorio; luego, este ADN modificado se introduce en un microorganismo (bacteria o levadura, generalmente) para que fabrique la proteína de interés; posteriormente, se selecciona aquella proteína que tenga la mejora deseada y esto se hace mediante mediciones analíticas precisas de la propiedad que te interesa: actividad enzimática, estabilidad en altas temperaturas, tolerancia a pH extremos u otras.
Las enzimas evolucionadas lo pueden hacer mejor
Le evolución molecular dirigida ha revolucionado la manera en que se mejoran las proteínas en el laboratorio y gracias a esto, la pionera Frances H. Arnold del California Institute of Technology, ha sido una de los científicos laureados con el Premio Nobel de Química este año. Ella y su grupo de investigación realizaron trabajos importantes con unas proteínas llamadas citocromos P450, las cuales se encuentran en muchos organismos. Los humanos tenemos varios citocromos P450 y participan en la transformación de compuestos extraños que ingresan en nuestro cuerpo. Esto lo realizan transfiriendo un átomo de oxígeno a las moléculas extrañas, lo cual las hace más fáciles de descomponer por otras enzimas o facilita su excreción. Esta capacidad catalítica extraordinaria se debe a que los citocromos tienen en su estructura un grupo químico llamado hemo, que contiene hierro. Este grupo hemo se encuentra en otro tipo de proteínas relacionadas, como la hemoglobina que transporta el oxígeno en nuestra sangre, las catalasas que descomponen el agua oxigenada y las hemo-peroxidasas de los hongos que participan en la degradación de la lignina, por mencionar algunas.
La doctora Arnold consiguió evolucionar al citocromo P450 de Bacillus megaterium para que pudiera catalizar una reacción llamada “ciclopropanación de alquenos”, una reacción muy socorrida en el área de síntesis orgánica para modificar dobles enlaces carbono-carbono (C=C). La reacción implica que la enzima debe transferir un átomo de carbono (en una forma activada llamada carbeno), en lugar de un átomo de oxígeno, como lo hace naturalmente. ¿Cómo se logró que la enzima cambiara drásticamente su actividad natural? Lo cierto es que la enzima sin ninguna modificación ya podía catalizar este tipo de reacción, pero como no era su actividad principal, esta era muy poco eficiente. Durante la evolución de los citocromos, quizá nunca estuvieron sometidos a la presión de selección para transferir carbonos, pero el potencial estaba allí. En el laboratorio, sí se sometieron a esta presión y es por eso que tras algunas generaciones se logró especializar a estas enzimas para esa tarea. La ventaja de esta enzima evolucionada con respecto a los métodos químicos conocidos hasta entonces es que la primera puede catalizar la reacción en un solo paso de reacción y a temperaturas moderadas; incluso la enzima podía funcionar dentro de las células bacterianas en las que se producía.
Evolución molecular dirigida en enzimas para generar una nueva química
Los experimentos de Arnold han demostrado que una función enzimática conocida (pero poco eficiente) se puede mejorar y adaptar a una cierta necesidad. Pero, ¿qué hay de reacciones químicas que naturalmente no se han descrito en los seres vivos pero que sí son usadas por los químicos en la industria? Los compuestos de carbono y silicio son muy utilizados en la industria química y de materiales. A pesar de que estos dos elementos son muy abundantes en la corteza terrestre, las enzimas no están adaptadas a catalizar reacciones que formen los enlaces carbono-silicio. La evolución no ha generado este tipo de enzimas, tal vez porque los seres vivos nunca se han enfrentado a la presión selectiva de formar estos enlaces químicos. Sin embargo, en el laboratorio se pueden exponer a las enzimas a este reto. La doctora Arnold también utilizó otra proteína llamada citocromo c de la bacteria Rhodothermus marinus para transferir los átomos de carbono a otros compuestos, y así lograr seleccionar la actividad de formación de enlaces carbono-silicio. Tras varias rondas de evolución dirigida, logró obtener una enzima evolucionada que era 15 veces más eficiente que los mejores métodos químicos sintéticos conocidos en ese momento. Además, la reacción se podía realizar a temperatura ambiente y con la enzima pura o incluso dentro de la bacteria que usó para expresarla. Este desarrollo abre la puerta para explorar el comportamiento de los seres vivos ante una química completamente nueva, lo cual podría tener importantes repercusiones en el diseño de moléculas con actividad biológica.
Virus convertidos en fábricas de proteínas
George P. Smith, de la Universidad de Missouri, fue otro de los galardonados gracias a que desarrolló un método conocido como el “despliegue de proteínas en fagos” (“phage display” en inglés), en el que se puede usar un bacteriófago, un virus que infecta a las bacterias, para desarrollar nuevas proteínas. Los virus son partículas infectivas relativamente simples, pues están formados básicamente por una molécula de ADN o ARN, que contiene la información genética para generar su envoltura o cápside de proteínas. Requieren infectar a una célula específica para poder aprovechar toda la maquinaria celular a su favor y así poder replicar su material genético y expresarlo para formar más proteínas de su cápside y finalmente generar muchas copias de sí mismos. Los fagos más utilizados en la técnica desarrollada por el doctor Smith son los llamados M13, fd, f1, T4 y λ. Los fagos más usados tienen forma de un bastón alargado, de 1 micra de largo y 0.006 micras de diámetro (1 micra es la milésima parte de un milímetro). Estos virus infectan a una cepa particular de la bacteria Escherichia coli que tiene la característica de presentar un apéndice en forma de hilo llamado el pilo F. Mediante la interacción del virus con este pilo el material genético desnudo (sin proteínas de la cápside) entra al citoplasma celular; las proteínas de la cápside quedan asociadas a la membrana plasmática. El ADN viral dentro de la célula bacteriana es replicado por las proteínas del hospedero; además, se transcribe y traduce la información genética para formar más proteínas de la cápside viral. Las nuevas moléculas del ADN viral son expulsadas de la célula y en el camino adquieren su cubierta proteica. Es interesante que las bacterias infectadas no mueren y, aunque crecen más lento, están secretando constantemente la progenie viral.
En la técnica de despliegue en fagos, se modifica el material genético del virus para que una proteína de interés se exprese fusionada a una de las proteínas que forman la cápside viral (Figura 2). Esto tiene una ventaja enorme, pues la proteína que queremos estudiar queda expuesta en la superficie del virus y en muchos casos se puede estudiar como si esta estuviera libre en la solución acuosa. Se puede crear una colección de fagos, que es una mezcla de muchos de estos virus que exhiben una proteína distinta en su superficie. Posteriormente se puede exponer esta mezcla a un sistema donde se encuentre una molécula específica inmovilizada en una superficie. Si la proteína exhibida en el fago interacciona con la molécula inmovilizada, esta quedará “capturada” con cierta fuerza. Se puede lavar la solución y quedarán unidas únicamente aquellas proteínas que se unieron con fuerza al ligante inmovilizado. Así, podemos ir seleccionando aquellas proteínas que tienen una alta afinidad por cierto ligante. Luego, infectamos nuevas bacterias con estos fagos seleccionados para que produzcan una mayor cantidad de ellos. Podemos realizar este procedimiento por varias rondas y en cada uno podemos introducir mutaciones al azar en el gen de interés e ir seleccionando en cada paso solo aquellas proteínas que muestran una mejoría. Al final, tendremos una proteína evolucionada, es decir, seleccionada y adaptada para nuestras propias necesidades.
El despliegue en fagos para la producción de anticuerpos terapéuticos
Sir Greg Winter, investigador del MRC Laboratory of Molecular Biology en Cambridge, Reino Unido, es el científico que cierra la terna de ganadores del Premio Nobel 2018 en Química. Gracias a su trabajo en el desarrollo de anticuerpos, más del 65% de los tratamientos terapéuticos en el mercado farmacéutico han sido posibles. Los anticuerpos son proteínas generadas por un tipo de célula muy especializada, llamada linfocito. Los linfocitos forman parte de nuestro sistema inmune y pueden producir a los anticuerpos, que son capaces de reconocer agentes extraños dentro del organismo.
Los estudios de Winter en anticuerpos de ratón utilizando la técnica de despliegue en fagos, para “humanizar” dichos anticuerpos, es la contribución que lo hizo ganador del premio nobel este año. Humanizar anticuerpos en este caso, significa que basándose en la secuencia de aminoácidos que conforman el anticuerpo generado por los linfocitos en el ratón, es posible cambiar dicha secuencia de aminoácidos, por una más parecida a la que tendrían los anticuerpos generados por los linfocitos humanos. Dado que los linfocitos pueden producir todo un repertorio de diferentes anticuerpos, es posible seleccionar aquellos que producen a los anticuerpos más específicos para atacar a algún agente extraño. Es posible aislar a dichas células, extraer la información genética para dicho anticuerpo y ponerla en el fago para su producción. A esto se le llama despliegue en fagos de anticuerpos monoclonales. La colección de anticuerpos puede ser evaluada y posteriormente probada en animales, como se explica en la Figura 3.
Con el tratamiento de anticuerpos monoclonales, ha sido posible tratar pacientes que padecen leucemia, un tipo de cáncer en la sangre y que comienza en la medula ósea. Estos nuevos tratamientos ya son aprobados por la Food and Drug Administration (FDA), que es la agencia norteamericana que regula los desarrollos farmacéuticos antes de ser puestos en el mercado. Con las terapias basadas en anticuerpos, es posible tener mejores resultados en los tratamientos contra el cáncer, dado lo específicos que resultan para atacar a las células cancerosas sin dañar a las células sanas, en comparación con el uso de medicamentos que, aunque matan a las células cancerosas, resultan muy tóxicos para el organismo y no son tan específicos. También se han empezado a utilizar estos tratamientos contra otros tipos de cáncer como el de pulmón o próstata, así como otras enfermedades como el asma.
En conclusión, los trabajos de estos investigadores nos han permitido usar el enorme potencial tanto de los catalizadores biológicos como de la producción de anticuerpos para satisfacer algunas de las necesidades de la química en la industria y la medicina. Incluso hemos podido crear “nueva química” utilizando el algoritmo de la evolución. Al final de cuentas, es la evolución la que ha dado origen a las formas más complejas y más bellas de la naturaleza.
Referencias
Jennifer Kan et al. Science 354 (2016) 1048-1051. http://science.sciencemag.org/content/354/6315/1048
Coelho et al. Science 339 (2013) 307-310.
http://science.sciencemag.org/content/339/6117/307
Smith, G.P., Petrenko, V.A., Chem. Rev. (1997) 97: 391-410.
https://pubs.acs.org/doi/abs/10.1021/cr960065d
Lecturas recomendadas
Ramírez Ramírez, J. y Ayala, M., 2014. “Enzimas: ¿qué son y cómo funcionan?”, Revista Digital Universitaria, No. 15, Vol. 12, pp. 1-13. ISSN: 1607-6079.
http://www.revista.unam.mx/vol.15/num12/art91/
Fuente: Academia de Ciencias de Morelos




